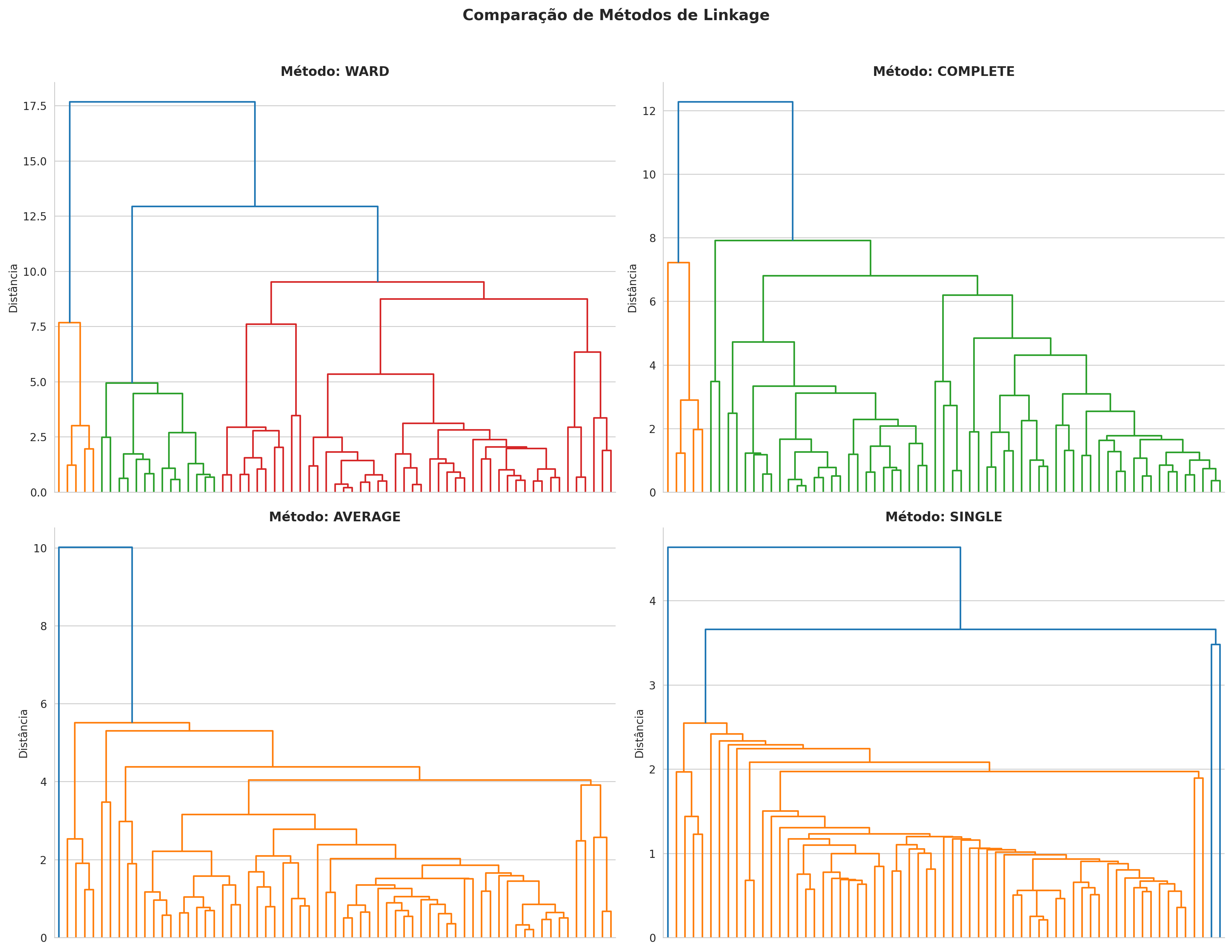
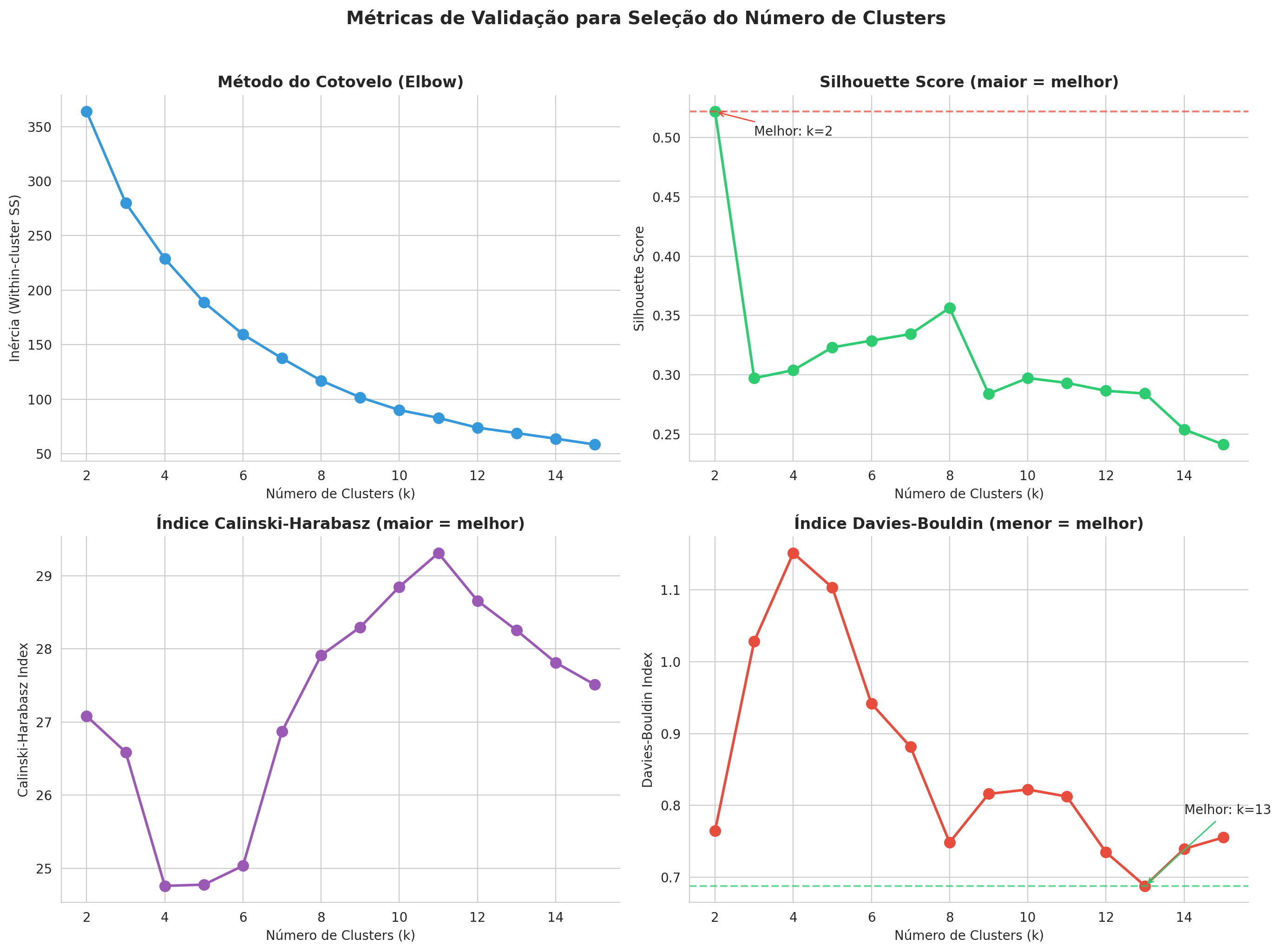
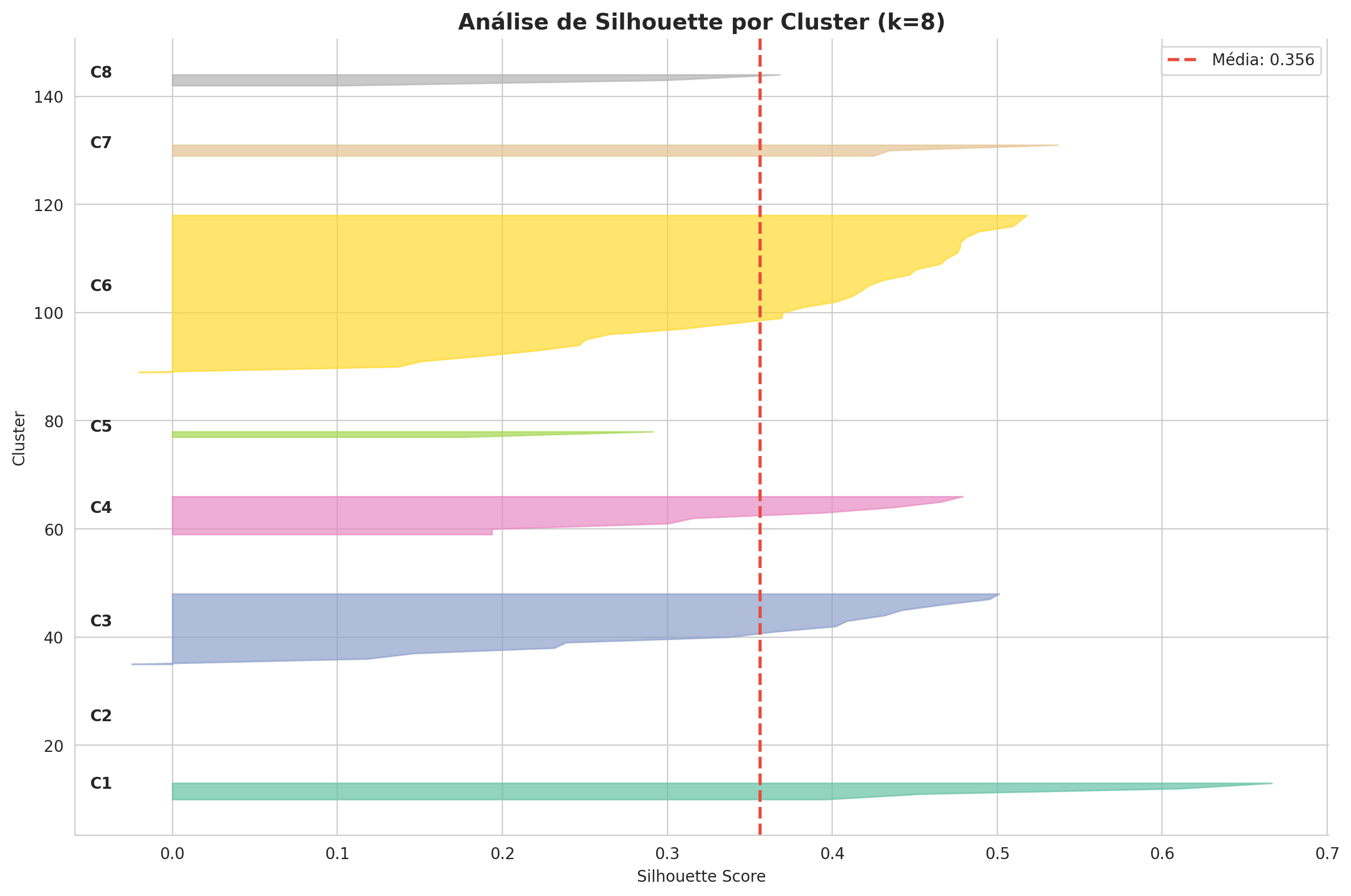
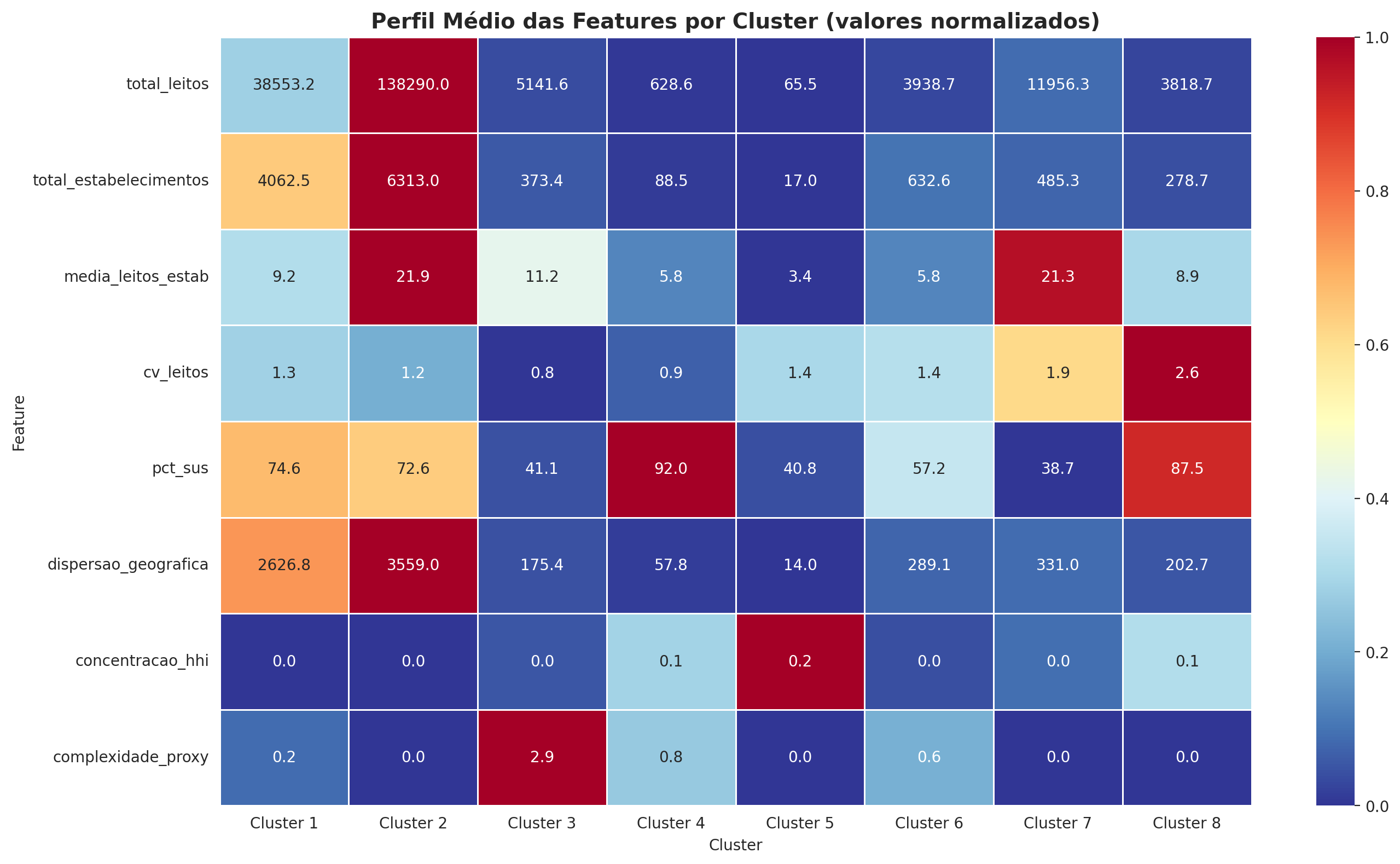
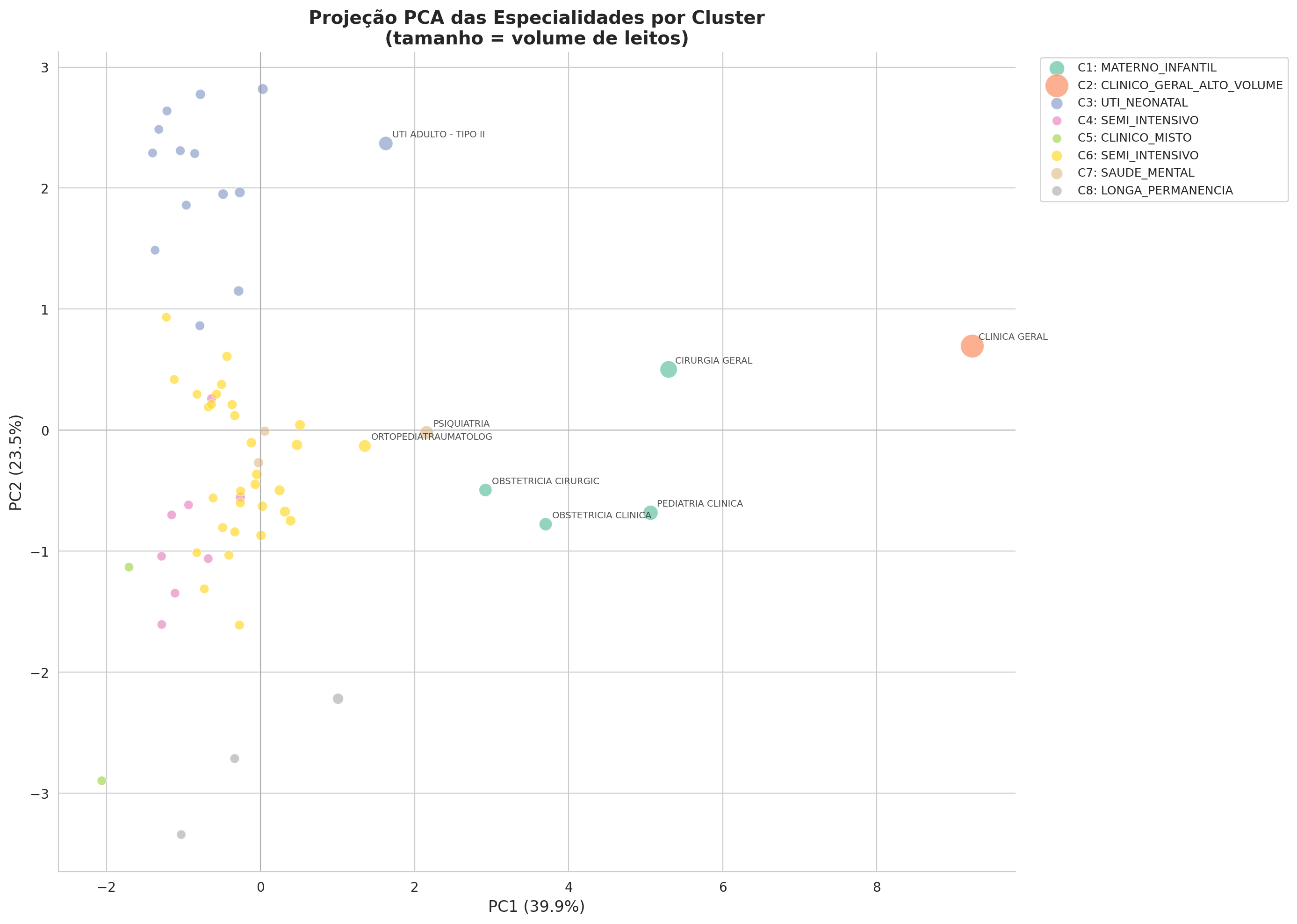
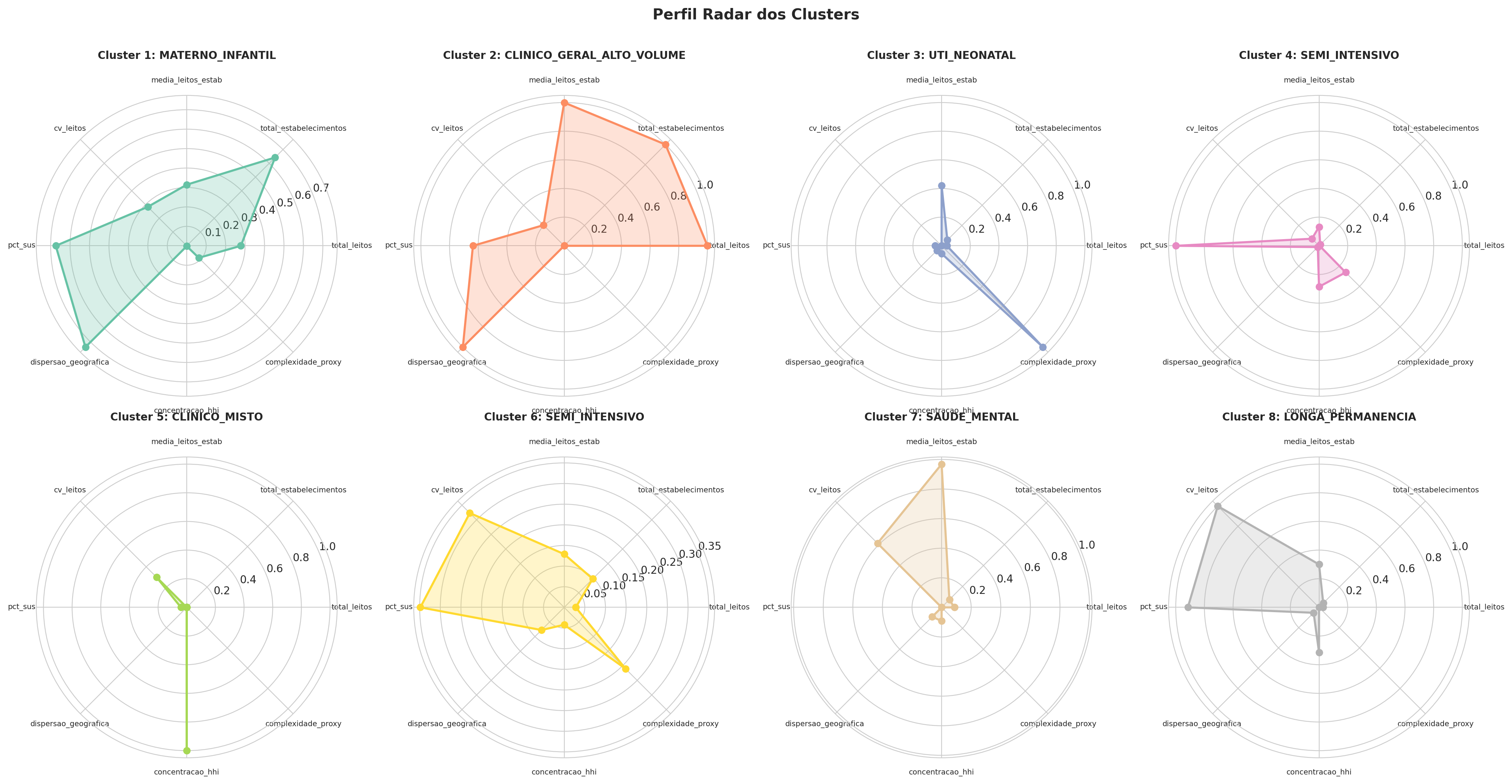
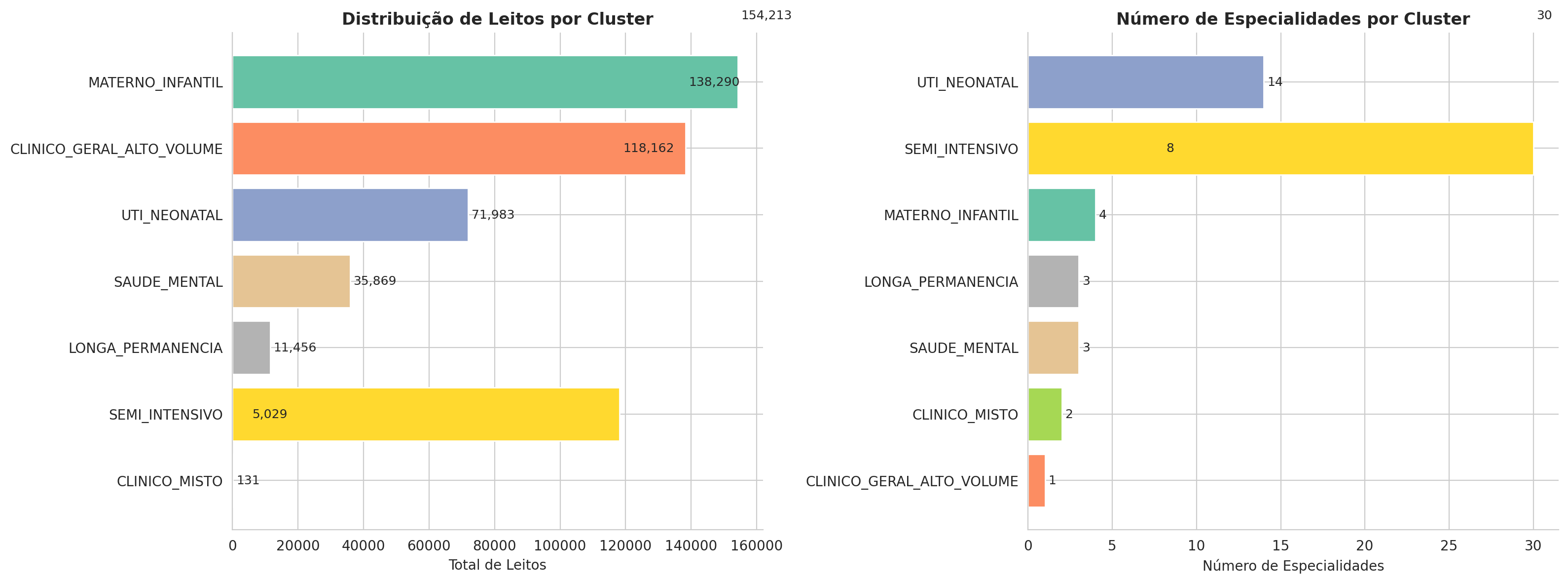
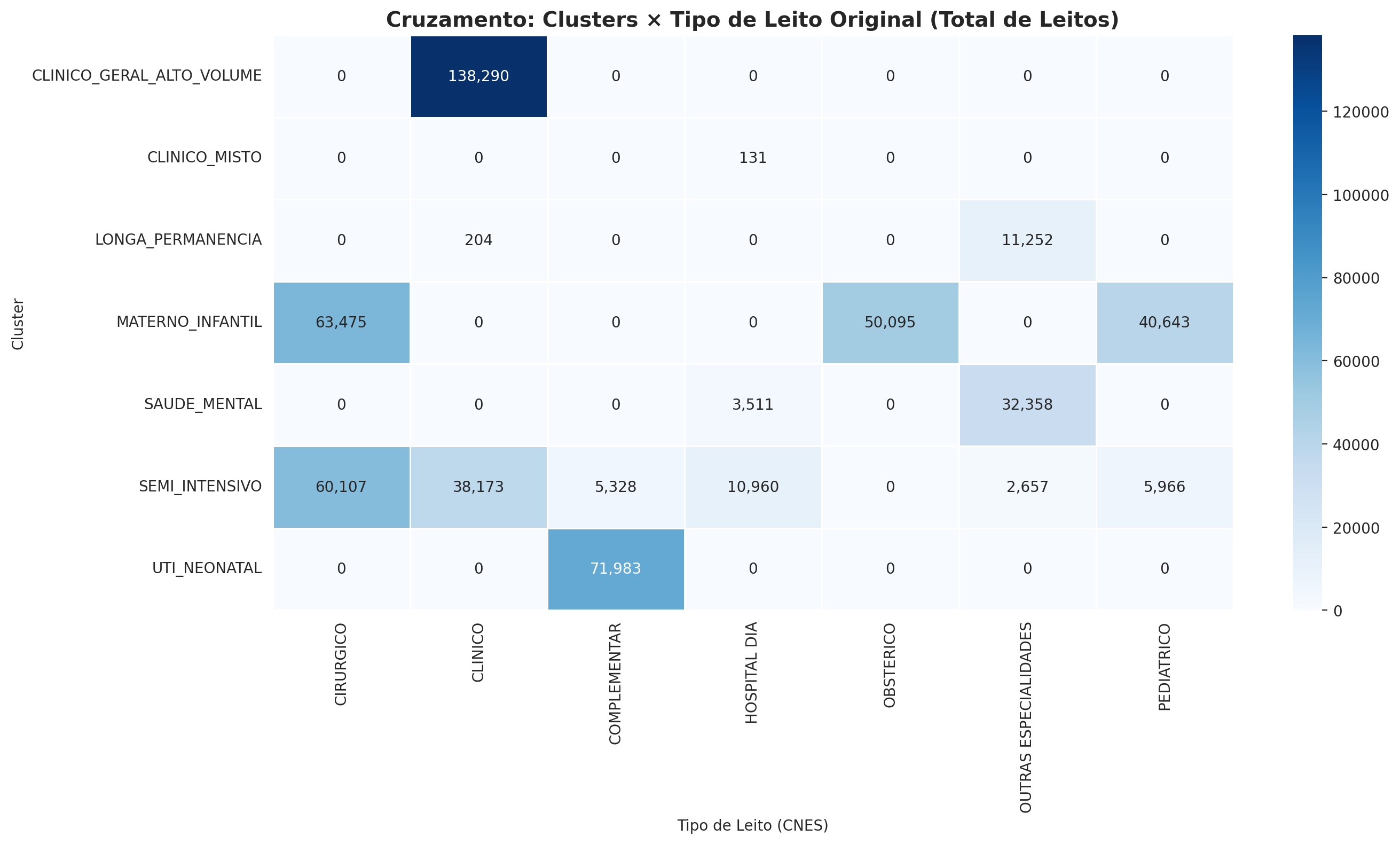
# Calcular inércia para diferentes valores de k
k_range = range(2, 16)
inertias = []
silhouettes = []
calinski = []
davies = []
for k in k_range:
# K-Means para inércia
kmeans = KMeans(n_clusters=k, random_state=42, n_init=10)
kmeans.fit(X_scaled)
inertias.append(kmeans.inertia_)
# Métricas de validação
labels_k = fcluster(Z, k, criterion='maxclust')
silhouettes.append(silhouette_score(X_scaled, labels_k))
calinski.append(calinski_harabasz_score(X_scaled, labels_k))
davies.append(davies_bouldin_score(X_scaled, labels_k))
# Visualização
fig, axes = plt.subplots(2, 2, figsize=(14, 10))
# Elbow Plot
axes[0, 0].plot(k_range, inertias, 'o-', color='#3498db', linewidth=2, markersize=8)
axes[0, 0].set_xlabel('Número de Clusters (k)')
axes[0, 0].set_ylabel('Inércia (Within-cluster SS)')
axes[0, 0].set_title('Método do Cotovelo (Elbow)', fontweight='bold')
axes[0, 0].spines['top'].set_visible(False)
axes[0, 0].spines['right'].set_visible(False)
# Silhouette Score
axes[0, 1].plot(k_range, silhouettes, 'o-', color='#2ecc71', linewidth=2, markersize=8)
axes[0, 1].set_xlabel('Número de Clusters (k)')
axes[0, 1].set_ylabel('Silhouette Score')
axes[0, 1].set_title('Silhouette Score (maior = melhor)', fontweight='bold')
axes[0, 1].axhline(y=max(silhouettes), color='#e74c3c', linestyle='--', alpha=0.7)
best_k_sil = list(k_range)[silhouettes.index(max(silhouettes))]
axes[0, 1].annotate(f'Melhor: k={best_k_sil}', xy=(best_k_sil, max(silhouettes)),
xytext=(best_k_sil+1, max(silhouettes)-0.02), fontsize=10,
arrowprops=dict(arrowstyle='->', color='#e74c3c'))
axes[0, 1].spines['top'].set_visible(False)
axes[0, 1].spines['right'].set_visible(False)
# Calinski-Harabasz
axes[1, 0].plot(k_range, calinski, 'o-', color='#9b59b6', linewidth=2, markersize=8)
axes[1, 0].set_xlabel('Número de Clusters (k)')
axes[1, 0].set_ylabel('Calinski-Harabasz Index')
axes[1, 0].set_title('Índice Calinski-Harabasz (maior = melhor)', fontweight='bold')
axes[1, 0].spines['top'].set_visible(False)
axes[1, 0].spines['right'].set_visible(False)
# Davies-Bouldin
axes[1, 1].plot(k_range, davies, 'o-', color='#e74c3c', linewidth=2, markersize=8)
axes[1, 1].set_xlabel('Número de Clusters (k)')
axes[1, 1].set_ylabel('Davies-Bouldin Index')
axes[1, 1].set_title('Índice Davies-Bouldin (menor = melhor)', fontweight='bold')
axes[1, 1].axhline(y=min(davies), color='#2ecc71', linestyle='--', alpha=0.7)
best_k_db = list(k_range)[davies.index(min(davies))]
axes[1, 1].annotate(f'Melhor: k={best_k_db}', xy=(best_k_db, min(davies)),
xytext=(best_k_db+1, min(davies)+0.1), fontsize=10,
arrowprops=dict(arrowstyle='->', color='#2ecc71'))
axes[1, 1].spines['top'].set_visible(False)
axes[1, 1].spines['right'].set_visible(False)
plt.suptitle('Métricas de Validação para Seleção do Número de Clusters', fontsize=14, fontweight='bold', y=1.02)
plt.tight_layout()
plt.show()
# Resumo
print("\nRESUMO DAS MÉTRICAS DE VALIDAÇÃO")
print("="*70)
metrics_df = pd.DataFrame({
'k': list(k_range),
'Silhouette': silhouettes,
'Calinski-Harabasz': calinski,
'Davies-Bouldin': davies
})
print(metrics_df.to_string(index=False))