Introdução
Este documento apresenta a criação de tipologias derivadas a partir dos dados de leitos hospitalares do CNES, utilizando quatro abordagens complementares:
Agrupamento Hierárquico (Tipo → Especialidade)Tipologia por Complexidade (UTI, Cirúrgico, Clínico)Tipologia por Público-Alvo (Adulto, Pediátrico, Obstétrico, Neonatal)Perfil de Estabelecimento (Porte × Natureza)
Ver código
import pandas as pdimport numpy as npimport warnings'ignore' )# Carregar dados tratados = pd.read_csv('arq2_tratado.csv' , sep= ';' , encoding= 'latin1' , low_memory= False )print (f"Registros: { len (df):,} " )print (f"Estabelecimentos: { df['cnes' ]. nunique():,} " )print (f"Leitos: { df['qt_exist' ]. sum ():,} " )
Registros: 49,804
Estabelecimentos: 9,072
Leitos: 535,133
Tipologia 1: Agrupamento Hierárquico
Estrutura de dois níveis: Tipo de Leito → Especialidade
Mapeamento Completo
Ver código
# Criar tipologia hierárquica 'TIPOLOGIA_HIERARQUICA' ] = df['DS_TP_LEITO' ] + ' > ' + df['DS_CO_LEITO' ]# Tabela hierárquica = []for tp in sorted (df['tp_leito' ].unique()):= df[df['tp_leito' ] == tp]['DS_TP_LEITO' ].iloc[0 ]= df[df['tp_leito' ] == tp].groupby(['co_leito' , 'DS_CO_LEITO' ])['qt_exist' ].sum ()= especialidades.reset_index().sort_values('qt_exist' , ascending= False )for _, row in especialidades.iterrows():'tp_leito' : tp,'Tipo' : tipo_nome,'co_leito' : row['co_leito' ],'Especialidade' : row['DS_CO_LEITO' ],'Leitos' : row['qt_exist' ]= pd.DataFrame(hierarquia)
0
1
CIRURGICO
3
CIRURGIA GERAL
63475
1
1
CIRURGICO
13
ORTOPEDIATRAUMATOLOGIA
20118
2
1
CIRURGICO
6
GINECOLOGIA
6877
3
1
CIRURGICO
2
CARDIOLOGIA
5368
4
1
CIRURGICO
12
ONCOLOGIA
5217
...
...
...
...
...
...
60
7
HOSPITAL DIA
73
SAUDE MENTAL
3511
61
7
HOSPITAL DIA
69
AIDS
445
62
7
HOSPITAL DIA
71
INTERCORRENCIA POS-TRANSPLANTE
385
63
7
HOSPITAL DIA
72
GERIATRIA
108
64
7
HOSPITAL DIA
70
FIBROSE CISTICA
23
65 rows × 5 columns
Resumo por Tipo de Leito
Ver código
= df.groupby('DS_TP_LEITO' ).agg({'co_leito' : 'nunique' ,'qt_exist' : 'sum' = {'co_leito' : 'Especialidades' , 'qt_exist' : 'Leitos' })'%' ] = (resumo_tipo['Leitos' ] / resumo_tipo['Leitos' ].sum () * 100 ).round (1 )= resumo_tipo.sort_values('Leitos' , ascending= False )
DS_TP_LEITO
CLINICO
15
176667
33.0
CIRURGICO
17
123582
23.1
COMPLEMENTAR
18
77311
14.4
OBSTERICO
2
50095
9.4
PEDIATRICO
2
46609
8.7
OUTRAS ESPECIALIDADES
5
46267
8.6
HOSPITAL DIA
6
14602
2.7
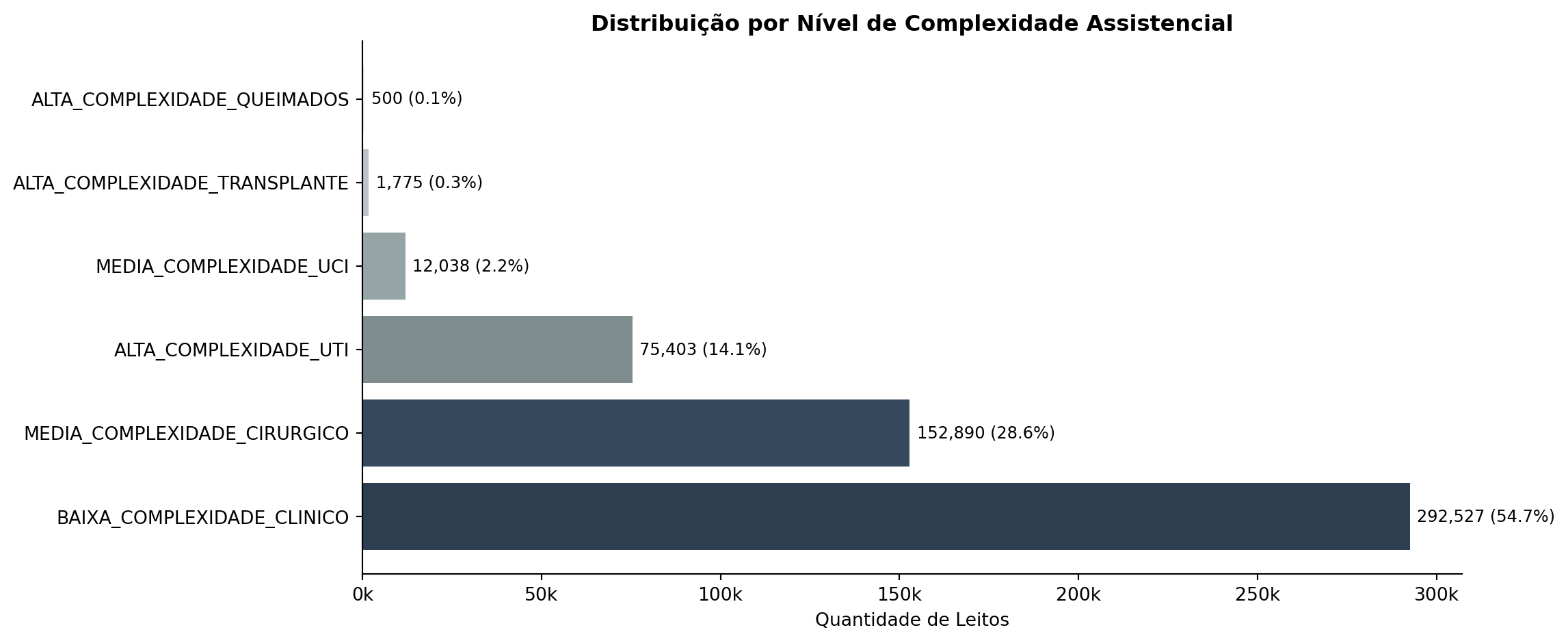
Tipologia 2: Por Complexidade
Classificação baseada na complexidade assistencial do leito.
Critérios de Classificação
ALTA_COMPLEXIDADE_UTI
UTI (todos os tipos)
ALTA_COMPLEXIDADE_QUEIMADOS
Leitos de queimados
ALTA_COMPLEXIDADE_TRANSPLANTE
Transplante
MEDIA_COMPLEXIDADE_UCI
Unidades de Cuidados Intermediários
MEDIA_COMPLEXIDADE_CIRURGICO
Leitos cirúrgicos
BAIXA_COMPLEXIDADE_CLINICO
Demais leitos clínicos
Ver código
def classificar_complexidade(row):= row['co_leito' ]= row['DS_CO_LEITO' ].upper() if pd.notna(row['DS_CO_LEITO' ]) else '' # UTI / Alta Complexidade if 'UTI' in ds or co in [74 ,75 ,76 ,77 ,78 ,79 ,80 ,81 ,82 ,83 ,85 ,86 ]:return 'ALTA_COMPLEXIDADE_UTI' # Unidades Intermediárias if 'INTERMEDIARI' in ds or 'CANGURU' in ds or co in [65 ,92 ,93 ,94 ,95 ,96 ]:return 'MEDIA_COMPLEXIDADE_UCI' # Queimados if 'QUEIMADO' in ds:return 'ALTA_COMPLEXIDADE_QUEIMADOS' # Transplante if 'TRANSPLANTE' in ds:return 'ALTA_COMPLEXIDADE_TRANSPLANTE' # Cirúrgico if row['tp_leito' ] == 1 or 'CIRURG' in ds:return 'MEDIA_COMPLEXIDADE_CIRURGICO' # Clínico return 'BAIXA_COMPLEXIDADE_CLINICO' 'TIPOLOGIA_COMPLEXIDADE' ] = df.apply (classificar_complexidade, axis= 1 )# Resumo = df.groupby('TIPOLOGIA_COMPLEXIDADE' )['qt_exist' ].agg(['count' , 'sum' ])= ['Registros' , 'Leitos' ]'%' ] = (tip_complex['Leitos' ] / tip_complex['Leitos' ].sum () * 100 ).round (1 )= tip_complex.sort_values('Leitos' , ascending= False )
TIPOLOGIA_COMPLEXIDADE
BAIXA_COMPLEXIDADE_CLINICO
23059
292527
54.7
MEDIA_COMPLEXIDADE_CIRURGICO
18971
152890
28.6
ALTA_COMPLEXIDADE_UTI
5835
75403
14.1
MEDIA_COMPLEXIDADE_UCI
1545
12038
2.2
ALTA_COMPLEXIDADE_TRANSPLANTE
289
1775
0.3
ALTA_COMPLEXIDADE_QUEIMADOS
105
500
0.1
Visualização
Ver código
import matplotlib.pyplot as plt= plt.subplots(figsize= (12 , 5 ))= ['#2c3e50' , '#34495e' , '#7f8c8d' , '#95a5a6' , '#bdc3c7' , '#ecf0f1' ]= ax.barh(tip_complex.index, tip_complex['Leitos' ], color= cores[:len (tip_complex)])'Quantidade de Leitos' , fontsize= 10 )'Distribuição por Nível de Complexidade Assistencial' , fontweight= 'bold' , fontsize= 12 )'top' ].set_visible(False )'right' ].set_visible(False )lambda x, p: f' { int (x/ 1000 )} k' ))for i, (v, pct) in enumerate (zip (tip_complex['Leitos' ], tip_complex['%' ])):+ 2000 , i, f' { v:,} ( { pct} %)' , va= 'center' , fontsize= 9 )
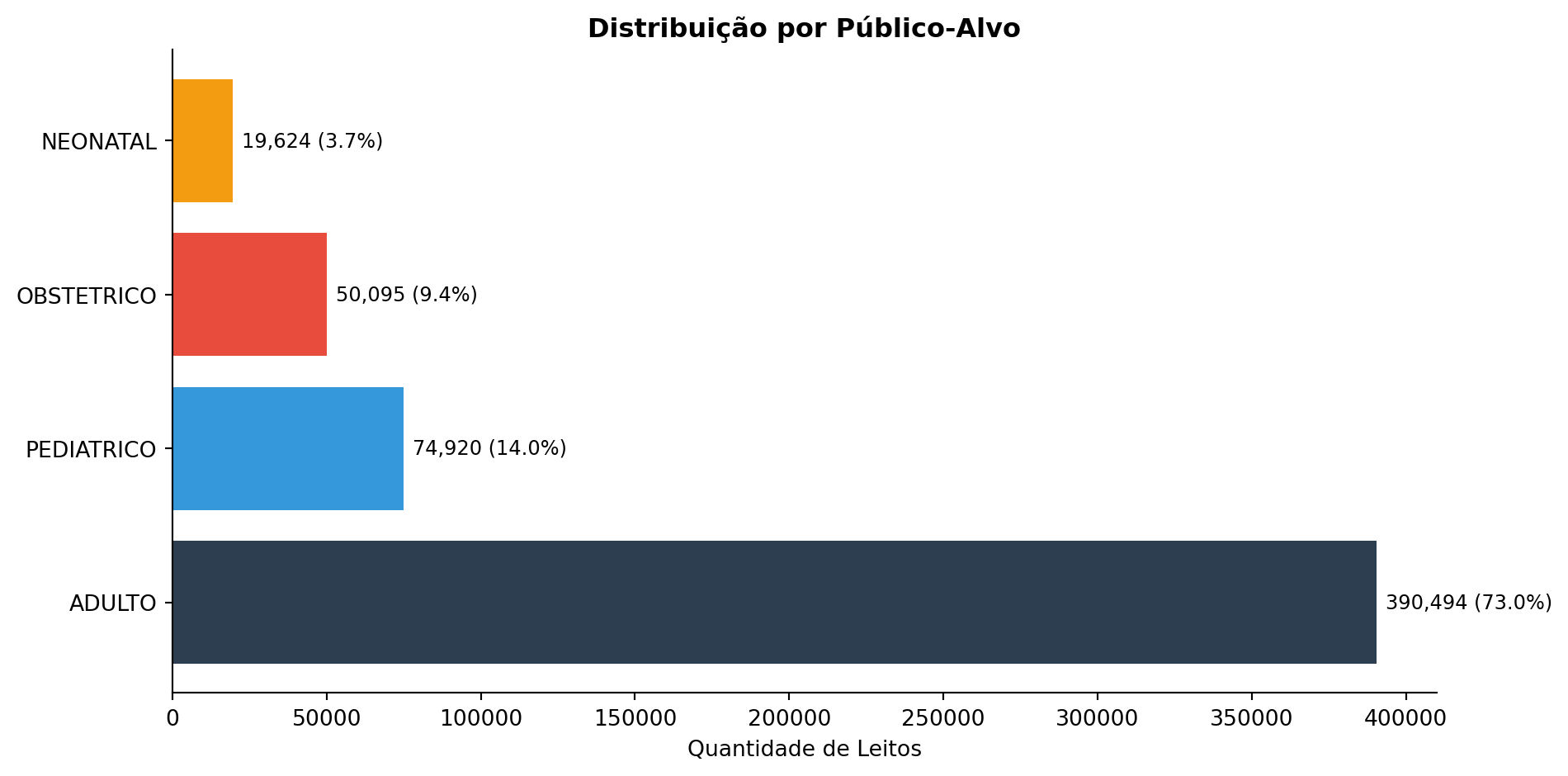
Tipologia 3: Por Público-Alvo
Classificação baseada no público atendido .
Critérios de Classificação
NEONATAL
Leitos neonatais e canguru
PEDIATRICO
Leitos pediátricos (tp_leito=5) ou com “PEDIATR” na descrição
OBSTETRICO
Leitos obstétricos (tp_leito=4)
ADULTO
Demais leitos
Ver código
def classificar_publico(row):= row['DS_CO_LEITO' ].upper() if pd.notna(row['DS_CO_LEITO' ]) else '' = row['tp_leito' ]# Neonatal if 'NEONAT' in ds or 'CANGURU' in ds:return 'NEONATAL' # Pediátrico if tp == 5 or 'PEDIATR' in ds:return 'PEDIATRICO' # Obstétrico if tp == 4 or 'OBSTETR' in ds:return 'OBSTETRICO' # Adulto return 'ADULTO' 'TIPOLOGIA_PUBLICO' ] = df.apply (classificar_publico, axis= 1 )# Resumo = df.groupby('TIPOLOGIA_PUBLICO' )['qt_exist' ].agg(['count' , 'sum' ])= ['Registros' , 'Leitos' ]'%' ] = (tip_publico['Leitos' ] / tip_publico['Leitos' ].sum () * 100 ).round (1 )= tip_publico.sort_values('Leitos' , ascending= False )
TIPOLOGIA_PUBLICO
ADULTO
32449
390494
73.0
PEDIATRICO
8448
74920
14.0
OBSTETRICO
6670
50095
9.4
NEONATAL
2237
19624
3.7
Visualização
Ver código
= plt.subplots(figsize= (10 , 5 ))= ['#2c3e50' , '#3498db' , '#e74c3c' , '#f39c12' ]= ax.barh(tip_publico.index, tip_publico['Leitos' ], color= cores_pub)'Quantidade de Leitos' )'Distribuição por Público-Alvo' , fontweight= 'bold' )'top' ].set_visible(False )'right' ].set_visible(False )for i, (v, pct) in enumerate (zip (tip_publico['Leitos' ], tip_publico['%' ])):+ 3000 , i, f' { v:,} ( { pct} %)' , va= 'center' , fontsize= 9 )
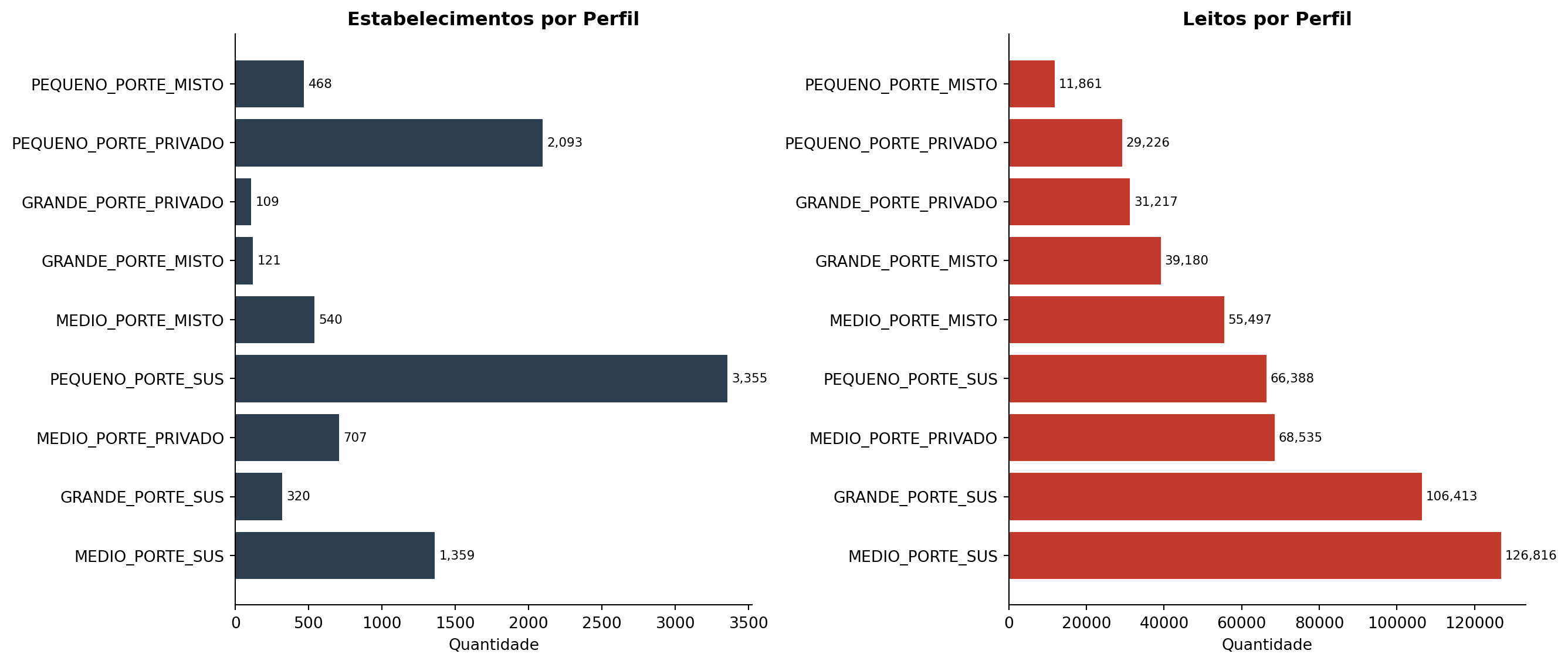
Tipologia 4: Perfil de Estabelecimento
Classificação dos estabelecimentos por Porte × Natureza (SUS/Privado) .
Critérios de Classificação
GRANDE_PORTE
≥ 200 leitos
MEDIO_PORTE
50-199 leitos
PEQUENO_PORTE
< 50 leitos
SUS
≥ 80% leitos SUS
PRIVADO
≤ 20% leitos SUS
MISTO
21-79% leitos SUS
Ver código
# Agregar por estabelecimento = df.groupby('cnes' ).agg({'qt_exist' : 'sum' ,'qt_sus' : 'sum' ,'qt_nsus' : 'sum' ,'tp_leito' : 'nunique' ,'co_leito' : 'nunique' ,'codufmun' : 'first' = ['cnes' , 'total_leitos' , 'leitos_sus' , 'leitos_nsus' , 'tipos_leito' , 'especialidades' , 'codufmun' ]'pct_sus' ] = (perfil['leitos_sus' ] / perfil['total_leitos' ] * 100 ).round (1 )# Classificação def classificar_perfil(row):= row['total_leitos' ]= row['pct_sus' ]# Por porte if leitos >= 200 := 'GRANDE_PORTE' elif leitos >= 50 := 'MEDIO_PORTE' else := 'PEQUENO_PORTE' # Por natureza if pct_sus >= 80 := 'SUS' elif pct_sus <= 20 := 'PRIVADO' else := 'MISTO' return f' { porte} _ { natureza} ' 'PERFIL_ESTABELECIMENTO' ] = perfil.apply (classificar_perfil, axis= 1 )# Resumo = perfil.groupby('PERFIL_ESTABELECIMENTO' ).agg({'cnes' : 'count' ,'total_leitos' : 'sum' ,'pct_sus' : 'mean' round (1 )= ['Estabelecimentos' , 'Leitos' , 'Media_SUS%' ]= resumo_perfil.sort_values('Leitos' , ascending= False )
PERFIL_ESTABELECIMENTO
MEDIO_PORTE_SUS
1359
126816
95.0
GRANDE_PORTE_SUS
320
106413
95.8
MEDIO_PORTE_PRIVADO
707
68535
0.8
PEQUENO_PORTE_SUS
3355
66388
98.8
MEDIO_PORTE_MISTO
540
55497
63.2
GRANDE_PORTE_MISTO
121
39180
62.3
GRANDE_PORTE_PRIVADO
109
31217
1.9
PEQUENO_PORTE_PRIVADO
2093
29226
0.2
PEQUENO_PORTE_MISTO
468
11861
60.3
Visualização
Ver código
= plt.subplots(1 , 2 , figsize= (14 , 6 ))# Por estabelecimentos = axes[0 ].barh(resumo_perfil.index, resumo_perfil['Estabelecimentos' ], color= '#2c3e50' )0 ].set_title('Estabelecimentos por Perfil' , fontweight= 'bold' , fontsize= 12 )0 ].set_xlabel('Quantidade' )0 ].spines['top' ].set_visible(False )0 ].spines['right' ].set_visible(False )for i, v in enumerate (resumo_perfil['Estabelecimentos' ]):0 ].text(v + 30 , i, f' { int (v):,} ' , va= 'center' , fontsize= 8 )# Por leitos = axes[1 ].barh(resumo_perfil.index, resumo_perfil['Leitos' ], color= '#c0392b' )1 ].set_title('Leitos por Perfil' , fontweight= 'bold' , fontsize= 12 )1 ].set_xlabel('Quantidade' )1 ].spines['top' ].set_visible(False )1 ].spines['right' ].set_visible(False )for i, v in enumerate (resumo_perfil['Leitos' ]):1 ].text(v + 1000 , i, f' { int (v):,} ' , va= 'center' , fontsize= 8 )
Resumo das Tipologias
Ver código
print ("=" * 70 )print ("RESUMO DAS TIPOLOGIAS CRIADAS" )print ("=" * 70 )print (" \n 1. TIPOLOGIA HIERÁRQUICA" )print (f" Níveis: 7 tipos → 65 especialidades" )print (f" Combinações únicas: { df['TIPOLOGIA_HIERARQUICA' ]. nunique()} " )print (" \n 2. TIPOLOGIA POR COMPLEXIDADE" )for tip in tip_complex.index:print (f" { tip} : { tip_complex. loc[tip, 'Leitos' ]:,} leitos ( { tip_complex. loc[tip, '%' ]} %)" )print (" \n 3. TIPOLOGIA POR PÚBLICO-ALVO" )for tip in tip_publico.index:print (f" { tip} : { tip_publico. loc[tip, 'Leitos' ]:,} leitos ( { tip_publico. loc[tip, '%' ]} %)" )print (" \n 4. PERFIL DE ESTABELECIMENTO" )for tip in resumo_perfil.index:print (f" { tip} : { int (resumo_perfil.loc[tip, 'Estabelecimentos' ]):,} estab. / { int (resumo_perfil.loc[tip, 'Leitos' ]):,} leitos" )
======================================================================
RESUMO DAS TIPOLOGIAS CRIADAS
======================================================================
1. TIPOLOGIA HIERÁRQUICA
Níveis: 7 tipos → 65 especialidades
Combinações únicas: 65
2. TIPOLOGIA POR COMPLEXIDADE
BAIXA_COMPLEXIDADE_CLINICO: 292,527 leitos (54.7%)
MEDIA_COMPLEXIDADE_CIRURGICO: 152,890 leitos (28.6%)
ALTA_COMPLEXIDADE_UTI: 75,403 leitos (14.1%)
MEDIA_COMPLEXIDADE_UCI: 12,038 leitos (2.2%)
ALTA_COMPLEXIDADE_TRANSPLANTE: 1,775 leitos (0.3%)
ALTA_COMPLEXIDADE_QUEIMADOS: 500 leitos (0.1%)
3. TIPOLOGIA POR PÚBLICO-ALVO
ADULTO: 390,494 leitos (73.0%)
PEDIATRICO: 74,920 leitos (14.0%)
OBSTETRICO: 50,095 leitos (9.4%)
NEONATAL: 19,624 leitos (3.7%)
4. PERFIL DE ESTABELECIMENTO
MEDIO_PORTE_SUS: 1,359 estab. / 126,816 leitos
GRANDE_PORTE_SUS: 320 estab. / 106,413 leitos
MEDIO_PORTE_PRIVADO: 707 estab. / 68,535 leitos
PEQUENO_PORTE_SUS: 3,355 estab. / 66,388 leitos
MEDIO_PORTE_MISTO: 540 estab. / 55,497 leitos
GRANDE_PORTE_MISTO: 121 estab. / 39,180 leitos
GRANDE_PORTE_PRIVADO: 109 estab. / 31,217 leitos
PEQUENO_PORTE_PRIVADO: 2,093 estab. / 29,226 leitos
PEQUENO_PORTE_MISTO: 468 estab. / 11,861 leitos
Exportação dos Dados
Ver código
# Salvar dataset com todas as tipologias = df.copy()'arq3_tipologias.csv' , sep= ';' , index= False , encoding= 'utf-8' )# Salvar perfil de estabelecimentos 'arq4_perfil_estabelecimentos.csv' , sep= ';' , index= False , encoding= 'utf-8' )print ("Arquivos exportados:" )print (" - arq3_tipologias.csv (leitos com tipologias)" )print (" - arq4_perfil_estabelecimentos.csv (perfil por CNES)" )
Arquivos exportados:
- arq3_tipologias.csv (leitos com tipologias)
- arq4_perfil_estabelecimentos.csv (perfil por CNES)
Dicionário das Tipologias
Tipologia por Complexidade
ALTA_COMPLEXIDADE_UTI
Unidades de Terapia Intensiva
UTI Adulto, Pediátrica, Neonatal, Coronariana, Queimados
ALTA_COMPLEXIDADE_QUEIMADOS
Leitos de Queimados
Queimado Adulto/Pediátrico (não UTI)
ALTA_COMPLEXIDADE_TRANSPLANTE
Transplantes
Intercorrência pós-transplante
MEDIA_COMPLEXIDADE_UCI
Unidades de Cuidados Intermediários
UCI Adulto, Pediátrico, Neonatal, Canguru
MEDIA_COMPLEXIDADE_CIRURGICO
Leitos Cirúrgicos
Tipo de leito = 1 (Cirúrgico)
BAIXA_COMPLEXIDADE_CLINICO
Leitos Clínicos
Demais leitos
Tipologia por Público-Alvo
ADULTO
Leitos para adultos
Padrão (não neonatal, pediátrico ou obstétrico)
PEDIATRICO
Leitos pediátricos
tp_leito=5 ou descrição contém “PEDIATR”
OBSTETRICO
Leitos obstétricos
tp_leito=4
NEONATAL
Leitos neonatais
Descrição contém “NEONAT” ou “CANGURU”
Perfil de Estabelecimento
GRANDE_PORTE_SUS
≥200 leitos
≥80% SUS
GRANDE_PORTE_MISTO
≥200 leitos
21-79% SUS
GRANDE_PORTE_PRIVADO
≥200 leitos
≤20% SUS
MEDIO_PORTE_SUS
50-199 leitos
≥80% SUS
MEDIO_PORTE_MISTO
50-199 leitos
21-79% SUS
MEDIO_PORTE_PRIVADO
50-199 leitos
≤20% SUS
PEQUENO_PORTE_SUS
<50 leitos
≥80% SUS
PEQUENO_PORTE_MISTO
<50 leitos
21-79% SUS
PEQUENO_PORTE_PRIVADO
<50 leitos
≤20% SUS
Elaborado por: Olavo CostaData: 21/01/2026Fonte: CNES - Competência 202506